기본개념: 코루틴, 메인루틴, 서브루틴
코루틴을 이해하기위해서는 메인루틴과 서브루틴을 이해하고 있으면, 더 쉽게 이해가 됩니다[이전포스팅]. 이 3가지 루틴을 정리하면 아래와 같습니다.
- 메인루틴(Main routine): 메인루틴은 보통 프로그램의 시작점이며, 프로그램의 주 흐름을 담당합니다. 메인루틴은 일련의 작업을 수행하고 다른 서브루틴이나 코루틴을 호출할 수 있습니다. 프로그램이 시작되는 메인코드라고 생각하면 됩니다.
- 서브루틴(subroutine): 메인루틴에서 호출되는 함수, 또는 서브루틴에서 호출되는 함수들을 의미합니다. 즉, 다른 루틴에서 호출되는 경우를 의미합니다.
- 코루틴(Coroutine): 메인루틴에서 호출되지만, 코루틴은 실행되는 도중에, 일시중단되어 다시 메인루틴으로 전환되었다가 다시 코루틴으러 전환될 수 있는 "제어흐름"이 가능한 루틴을 의미합니다.
코루틴과 제너레이터
코루틴과 제너레이터 중, 코루틴이 더 일반적인 개념이며 코루틴 중에 반복가능한 데이터를생성하는 한 종류가 제너레이터라고 할 수 있습니다. 이 제너레이터와 코루틴을 굳이 비교하자면 아래와 같이 비교해볼 수 있습니다.
| 코루틴 | 제너레이터 | |
| 공통점 | 1. 함수 형태로 작성 2. yield 키워드로 생성하거나 반환 3. 상태유지: 이전 상태를 유지하며, 중단시점부터 재개가 가능 |
1. 함수 형태로 작성 2. yield 키워드로 생성하거나 반환 3. 상태유지: 이전 상태를 유지하며, 중단시점부터 재개가 가능 |
| 종료 | yield 완료시: StopIteration예외 | yield 완료시: StopIteration예외 |
| 차이점 | ||
| 목적 | 주로 비통기 작버을 처리하기 위해 사용 | 반복 가능한 객체를 사용 예) yield 1; yield 2.. |
| 작성방법 | yield 외에 await을 이용해서 중간에 값을 보내거 받을 수 있음 | |
| 호출방법 | send()메서드로 값을 주고 받음. | next()함수로 값을 하나씩받음 |
| 종료 | StopIteration 예외 | StopIteration 예외 |
코루틴의 간단한 예시입니다.
def simple_coroutine():
print("Coroutine started")
x = yield # 처음 호출될 때까지 대기
print(f"Received: {x}")
y = yield x * 2 # yield 우측에 있는 값은 반환하는 값입니다.
print(f"Received: {y}")
# 코루틴 생성
coro = simple_coroutine()
# 코루틴 시작
print(next(coro)) # Coroutine started, 첫 번째 yield까지 실행
# 데이터 전송
print(coro.send(10)) # Received: 10
# 데이터 전송
coro.send(20) # Received: 20
위의 예시처럼 코루틴을 시작하려면 반드시 "next()"을 호출하여 실행시켜야 합니다. 그렇지 않으면 아래와 같이 에러가 발생합니다. 즉, coroutine 시작지점까지는 함수가 실행되어 대기중이어야합니다.
Cell In[24], line 1
----> 1 coro.send(20)
TypeError: can't send non-None value to a just-started generator
코루틴과 마찬가지로, generator도 yield의 반환이 없으면 StopIteration 예외가 발생합니다.
In [1]: def simple_generator():
...: yield 1
...: yield 2
...: yield 3
...:
In [2]: gen = simple_generator()
In [4]: next(gen)
Out[4]: 1
In [5]: next(gen)
Out[5]: 2
In [6]: next(gen)
Out[6]: 3
In [7]: next(gen)
StopIteration Traceback (most recent call last)
Cell In[7], line 1
----> 1 next(gen)
StopIteration:
코루틴의 4가지 상태: GEN_CREATE, GEN_RUNNING, GEN_SUSPENDED, GEN_CLOSE
- GEN_CREATED (생성 상태): 코루틴이 생성되었지만 아직 시작되지 않은 상태입니다.
- 특징: __next__() 또는 send(None)이 호출되기 전의 상태입니다.
def coroutine_example():
yield
coro = coroutine_example()
print(coro) # 코루틴 객체가 생성된 상태, 아직 실행되지 않음2. GEN_RUNNING (실행 중 상태): 코루틴이 현재 실행 중인 상태입니다.
- 특징: 코루틴이 실행되고 있으며, 내부적으로 yield 문을 만날 때까지 실행됩니다. 실행 중에 다시 재개될 수 없습니다.
def coroutine_example():
print("Running")
yield
coro = coroutine_example()
next(coro) # "Running" 출력, 실행 중 상태
3. GEN_SUSPENDED (일시 중지 상태): 코루틴이 yield 문에서 일시 중지된 상태입니다. 주의해야할 것은 RUNNING 상태는 yield을 만나기 전까지 실행되는 것인데, 여기서는 yield가 이미 실행된 상태를 의미합니다.
그리고, inpsect.getgeneratorstate 함수로 코루틴의 상태를 조회해보면 GEN_SUSPENDED을 확인할 수 있습니다.
>>> def coroutine_example():
>>> yield "Suspended"
>>>
>>> coro = coroutine_example()
>>> print(next(coro))
>>> import inspect
>>> inspect.getgeneratorstate(coro)
>>> 'GEN_SUSPENDED'
4. GEN_CLOSED(종료상태): 코루틴이 종료된 상태입니다. 종료되면 다시 시작할 수 없습니다.
- 특징: 모든 코드가 실행되었거나 close() 메서드가 호출된 상태입니다.
def coroutine_example():
yield "Running"
return "Done"
coro = coroutine_example()
print(next(coro)) # "Running" 출력
try:
next(coro) # StopIteration 예외 발생
except StopIteration as e:
print(e.value) # "Done" 출력, 종료 상태
각 상태를 연속선상으로 확인해보면 아래처럼 확인해볼 수 있습니다.
import inspect
def coroutine_example():
print("Coroutine started")
x = yield "Suspended at first yield" # GEN_SUSPENDED 상태로 진입
print(f"Received: {x}")
y = yield "Suspended at second yield" # 다시 GEN_SUSPENDED 상태로 진입
print(f"Received: {y}")
return "Done" # GEN_CLOSED 상태로 진입
# 코루틴 객체 생성 (GEN_CREATED 상태)
coro = coroutine_example()
print(inspect.getgeneratorstate(coro)) # GEN_CREATED
# 코루틴 시작 (GEN_RUNNING 상태)
print(next(coro)) # "Coroutine started" 출력, "Suspended at first yield" 반환
print(inspect.getgeneratorstate(coro)) # GEN_SUSPENDED
# 데이터 전송 및 재개 (GEN_RUNNING 상태로 변환 후 GEN_SUSPENDED 상태로 다시 변환)
print(coro.send(10)) # "Received: 10" 출력, "Suspended at second yield" 반환
print(inspect.getgeneratorstate(coro)) # GEN_SUSPENDED
# 데이터 전송 및 종료 (GEN_RUNNING 상태로 변환 후 GEN_CLOSED 상태로 변환)
try:
coro.send(20) # "Received: 20" 출력, StopIteration 예외 발생
except StopIteration as e:
print(e.value) # "Done" 출력
print(inspect.getgeneratorstate(coro)) # GEN_CLOSED
while True 구문을 이용한 코루틴
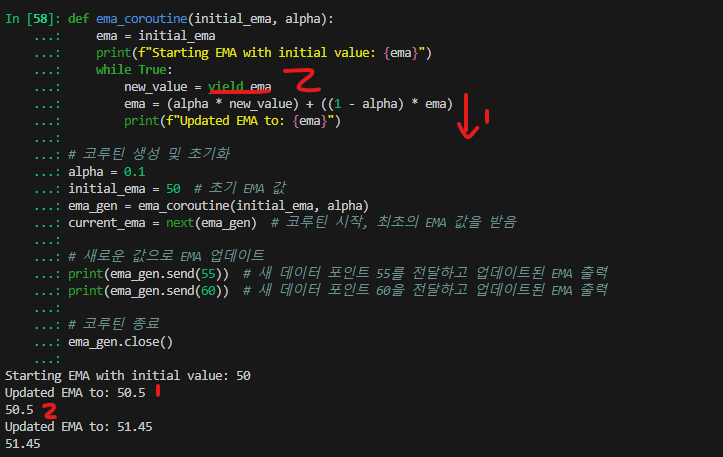
while True를 사용한 코루틴으로 지수 이동 평균 (Exponential Moving Average, EMA)을 계산하는 것들을 해볼 수 있습니다.
아래는 그 예제입니다.
def ema_coroutine(initial_ema, alpha):
ema = initial_ema
print(f"Starting EMA with initial value: {ema}")
while True:
new_value = yield ema
ema = (alpha * new_value) + ((1 - alpha) * ema)
print(f"Updated EMA to: {ema}")
# 코루틴 생성 및 초기화
alpha = 0.1
initial_ema = 50 # 초기 EMA 값
ema_gen = ema_coroutine(initial_ema, alpha)
current_ema = next(ema_gen) # 코루틴 시작, 최초의 EMA 값을 받음
# 새로운 값으로 EMA 업데이트
print(ema_gen.send(55)) # 새 데이터 포인트 55를 전달하고 업데이트된 EMA 출력
print(ema_gen.send(60)) # 새 데이터 포인트 60을 전달하고 업데이트된 EMA 출력
# 코루틴 종료
ema_gen.close()
yield 에서 값을 반환하는 순서를 주의할 필요가 있습니다.
- send(50): 코루틴에 값을 보내면 new_value에 할당합니다. 이후 print()문까지 도달하여 출력합니다.
- yield ema: 그리고나서 업데이트된 ema을 반환하여 50.5가 출력됩니다.
코루틴을 초기화해주면 데코레이터: coroutine
'Data science > Python' 카테고리의 다른 글
| [pytorch] register_buffer 설명 및 사용 방법 (0) | 2024.09.27 |
|---|---|
| numpy array의 stride란? (0) | 2024.07.10 |
| python with 구문 & context manager (1) | 2024.06.19 |
| python 매직메서드 (__repr__, __str__, __slots__) (0) | 2024.06.03 |
| 파이썬 바이트 표현 (bytes, bytearray) (0) | 2024.05.07 |