Preliminary
- Yottixel: 병리이미지 검색(슬라이드 단위) RGB히스토그램 기반 패치 클러스터링 방법. 패치를 뽑는 과정에서 작위적일 수 있다는 단점이 있음.
Motivation
- 데이터베이스 구축(index dataset=Atlas)
- 병리의사는 normal histology에 대해서 정확히 알고 있어서, 병리적인 판단할 때 정상부분은 무시하고 비정상 부분에 대해서 집중함.
- 이것을 AI로 구현하려면 normal tissue인 패치를 구분
Methods: One-class classifier
dataset
- cSCC(cutaneous squamous cell carcinoma): 660 슬라이드
- Breast tissue: 21WSIs, 432 WSIs(for validation)
Normal altas 생성: outlier detection (One-class classifier)
- Isolation Forest: Tree기반에서 분기를 잘 타는 경우(in-class data), 트리에서 분기를 잘 안타는 경우(outerlier)
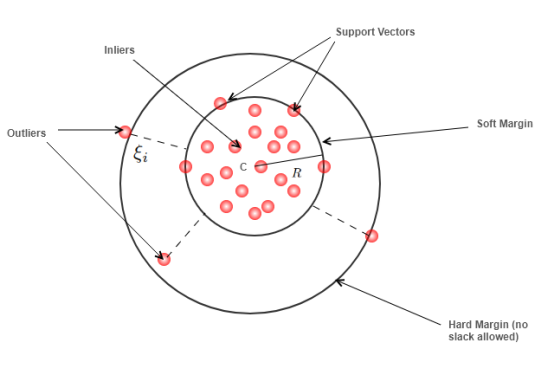
- one-class SVM: 정상데이터만 학습하는 비지도학습 방법.
- Objective function: $min_{w, \rho ,\epsilon} \frac{1}{2} ||w||^{2} + \frac{1}{vn} \sum_{i=1}^{n}(\epsilon_{i}-\rho) $
- $f(x)=w^{T}\Phi(x) - \rho$: 가 고차원공간으로 매핑하는 함수
- $f(x) >= 0 $ 이면 정상, $f(x)<0$이면 비정상으로 만듬
- 즉, 정상데이터는 c데이터 중심으로 있기를 원함.
- $v$: 마진에러를 고려. WSIs의 아티팩
- Objective function: $min_{w, \rho ,\epsilon} \frac{1}{2} ||w||^{2} + \frac{1}{vn} \sum_{i=1}^{n}(\epsilon_{i}-\rho) $
데이터 전처리: Unet 기반 tissue detecton -> patchification (20x) -> feature extractor 뽑음(DenseNet121, DINO, resnet50)
검색
- 클러스터링: WSI가 주어지면, RGB값 기반으로 9개의 클러스터링을 진행
- 모자이킹: 각 클러스터에서 공간을 고려하여 다양하게 15%의 패치 선택함 (모자이크)
- 패치화: 특징화 진행. query feature vectors (N개)가 획득
- DB검색:
- DB에 있는 다른 WSI(M개 패치)와 one-to-one 거리비교: query feature x WSI(in DB) feature 비교
- 거리비교한 1/2*N*M에서 에서 중각값을 최종 거리로 사용.
- 진단명 생성: 유사도 높은 Top N개에서 Majority votting으로 진단명생성
https://www.nature.com/articles/s41598-024-54489-9#Sec2

반응형



