1. 데이터 소스
- 다양한 염색, 스캐너, 생물학적 오브젝트의 데이터셋을 구성했습니다. 공공데이터 뿐만아니라, 따로(Private)모은 데이터셋을 포함해서 총 158,852 Slide이미지를 구했습니다. 이 약 16만장의 이미지를 4개 종류의 Resolution으로 1억 9천만장의 패치를 얻었습니다.
- 장기 수: 50개 이상
- 질환 그룹: 28종류 이상
- 스캐너: 11종류
- 배율: 20x, 40x 각
- 조직 영역 검출(Tissue regions): ArtifactDetection라는 PathAI의 Fore/Background 이미지를 분리하는 로직을 넣었습니다. 전경만 뽑았구요. 외부로 공개되지 않았지만 오츄알고리즘 보다, 다양한 상황(스캐너/염색) 등에서 더 좋은 성능을 보인다고합니다.
- 타일링(Tiling): 40x (0.25mpp), 20x (0.5mpp), 10x(1mpp), 5x (2mpp)로 배율로 위에서 얻은 전경의 티슈만 뽑았습니다.
2. 학습을 위한 구성요소(아키텍처)
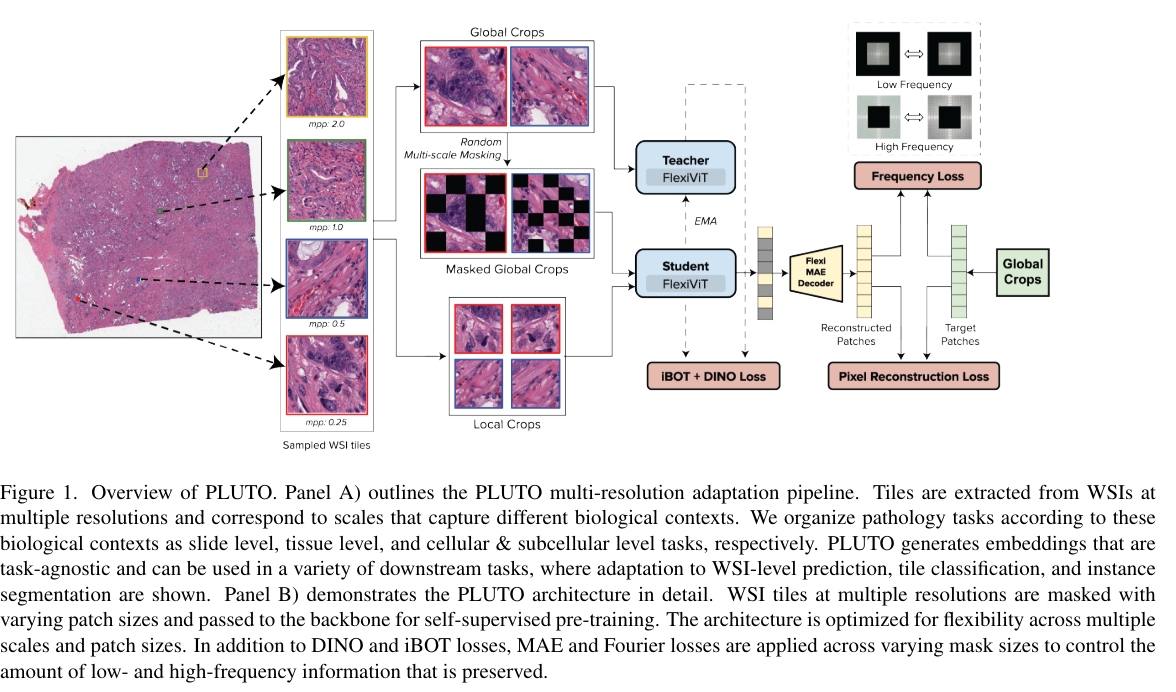
PLUTO은 DINO v2의 학습방법을 기본적으로 따라갑니다. DINO v2은 DINO라는 라벨링 없이 하는 지식증류방법과 iBOT이라는 MIM(Maksed image modeling)방법으로 학습하는 방법입니다(URL).
DINO v2은 자연계에서 얻어진 이미지들이고, PULTO은 병리이미지를 위한 모델이니 이 차이점을 반영합니다. WSI에는 핵, 세포, 분비샘(gland)등 이 다양한 사이즈에서 관찰되니, 이를 더 잘 반영할 수 있도록(glanularity), MAE(Masked autoencoder)을 추가합니다.
이 아키텍처 및 학습방법에 들어간 기술적인 방법은 아래와 같습니다.
- DINO v2
- Masked AutoEncoder(MAE)
- FlexiViT scheme
- Fourier loss based term
이 중, 2, 3, 4번에 해당하는 내용만 서술해보겠습니다. DINO v2은 여기를 참고하세요
2. MAE(Masked AutoEncoder) objective: 입력이미지의 일부를 재구성하도록 합니다. MAE은 학습에 원본이미지의 다수(예 75%)을 마스킹하고, 인코딩한 결과를 각각 벡터로 얻고, 이 벡터로 이미지를 복원하는 연산을하는 디코더가 한 묶음입니다. 이 복원은 maksed patch로 픽셀을 만든 것만 MSE의 목적함수를 갖습니다 [URL]. 일반적인 오토인코더와의 차이점은 1)ViT을 이용하면서 더 작은 단위의 패치이미지로 재구성한다는 것, 2)마스킹 했다는 것이 차이점입니다.

또한, 패치사이즈를 유연하게 받을 수 있게, FlexiViT을 사용합니다. FlexViT을 이용하는 경우 병리이미지의 배율을 조정하여 학습하기 있기 때문에 사용합니다. FlexiViT의 기술적 설명 아래와 같습니다.
3. FlexiViT: 보통 Vision Transformer은 패치 크기를 낮추게되면서 패치수가 증가하는데 성능은 좋아지지만 컴퓨팅비용은 커집니다. FlexiVIt은 학습시에 랜덤하게 패치 사이즈를 조절해가면서(=token 수가 증가하거나 감소하면서) 학습하는 방법을 의미합니다.

4. Fourier-loss based term: 푸리에 관련 손실 함수를 목적함수에 추가했다고 합니다. 이 손실함수에 대해서는 명확히 설명/참조가 되어있지않지만, 이미지를 푸리에변환 후(URL), 복원한 푸리에주파수와 원본 푸리에주파수를 비교한 것처럼 보입니다.
3. 학습 방법 (구현)
PLUTO은 전체적으로 DINO v2의 아키텍처를 따릅니다. 자세한 순서는 아래와 같습니다.
- 샘플링: 여러 Resolution으로 타일링한 이미지에서 1장을 뽑습니다.
- Cropping: 이 한장으로 2장의 이미지를 만듭니다. 하나는 224 crop하여 global view을 만들고, 또 하나는 96 size로 만들어 local view로 만듭니다.
- Masking & Forwarding:
- forwarding: local view은 student 모델에 들어가 포워딩되고, global view은 teacher model에 포워딩됩니다. 당연히 DINO v2의 iBOT 구성을 따르기에 student model에 들어가는 crop view도 일부를 마스킹합니다. 두 모델은 FlexiViT입니다.
- parameter update: DINO v2와 같이 Teacher 모델의 파라미터는 student model의 파라미터를 EMA(Exponential moving average)을 한 후 업데이트하도록함.
- global view masking: global view에 해당하는 부분은 마스킹을 하여 FlexiVIT에 넣습니다.
- MAE decoder: student model에서 나온 임베딩값들을 MAE decoder모델에 넣어 적용합니다. 이 MAE에서 나온 masked patch embeddign 값은 global view의 patch 픽셀들과 비교하게됩니다(L2 loss).
- Fourier transform loss: 재구축한 푸리에변환 주파수와 global view의 푸리에주파수를 비교합니다. L2 loss 입니다.
요약하면 총 손실함수는 $L= L_{DINO} + L_{iBOT} + L_{MAE} + L_{Fourier}$ 으로 구성됩니다.
Slide level prediction
Slide level prediction은 addictive MIL을 이용했습니다. Addictive MIL은 PathAI에서 고안한 MIL방법론이며, 사후해석이 아니라 모델 자체에서 해석이 가능하도록 집계하는 방식입니다(URL).
$p_{Addictive}(x)=\sum_{i=1}^{N}\psi_{p} (m_{i}(x))$
- $ \psi_{p} (m_{i}(x))$: $i$패치에서 class-wise contibution을 계산합니다. 패치수가 N개고 클레수가 C개라면 $\mathbb{R}^{C}\timesN$의 메트릭스를 생산합니다. 이후 N축(axis=1)으로 합하여 C만남겨, confidence을 계산합니다.
이 Addictive MIL에서는 이론적인 설명이 매우 잘되어있습니다.

